2019 9/8 インストール追記
公式サイト
http://qualimap.bioinfo.cipf.es
ユーザーマニュアル
http://qualimap.bioinfo.cipf.es/doc_html/index.html
ワークフロー
http://qualimap.bioinfo.cipf.es/doc_html/workflow.html
CUI環境でのラン。
http://qualimap.bioinfo.cipf.es/doc_html/command_line.html
インストール
依存
- Picard: a Java API (SAM-JDK) for creating programs that read and write SAM files.
- FastQC: a quality control tool for high throughput sequence data.
- SAMTools: essential utilities for manipulating alignments in the SAM format.
- NOISeq (リンク) quality control and differential gene expression analysis for RNA-seq data.
- Repitools (リンク)quality assessment, visualization, summarization and statistical analysis of epigenomics experiments.
公式サイトからダウンロードしてフォルダにパスを通す。
#解凍して中に入り、次のように打つ。
echo export PATH=\$PATH:`pwd`\ >> ~/.bash_profile && source ~/.bash_profile
#またはcondaを使う、ここではcondaの仮想環境に導入。
#biocona (link)
conda create -n qualimap-env -y qualimap
conda activate qualimap-env
> qualimap -h
$ qualimap -h
Java memory size is set to 1200M
Launching application...
Java HotSpot(TM) 64-Bit Server VM warning: ignoring option MaxPermSize=1024m; support was removed in 8.0
QualiMap v.2.2.1
Built on 2016-10-03 18:14
usage: qualimap <tool> [options]
To launch GUI leave <tool> empty.
Available tools:
bamqc Evaluate NGS mapping to a reference genome
rnaseq Evaluate RNA-seq alignment data
counts Counts data analysis (further RNA-seq data evaluation)
multi-bamqc Compare QC reports from multiple NGS mappings
clustering Cluster epigenomic signals
comp-counts Compute feature counts
Special arguments:
--java-mem-size Use this argument to set Java memory heap size. Example:
qualimap bamqc -bam very_large_alignment.bam --java-mem-size=4G
ラン
qualimap --java-mem-size=4G #GUI版が開く
File -> New analysis -> BAM QCを選択。

BAMファイルを選択(SAMも可能だがposition sort されている必要がある)。

>>> Start analysisをクリックして解析開始。
マッピングの詳細。

coverage

coverage histgram

duplication率

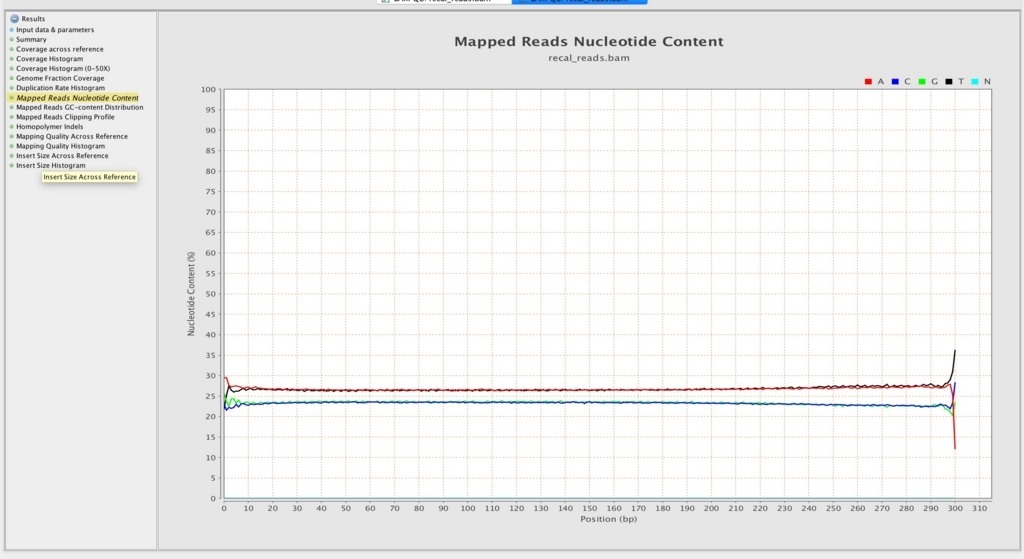
リードのポジション毎の塩基の分布
GC率の分布

リードのポジション毎のクリッピング率

Homo polymer

Mapping quality

Mapping qualityのヒストグラム

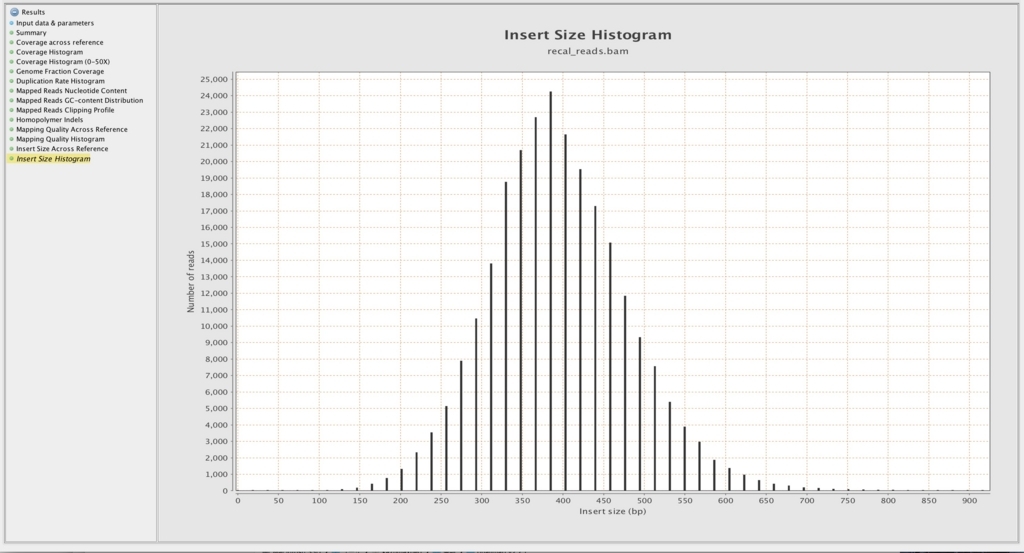
インサートサイズ

インサートサイズの分布
結果はexport as PDFまたはHTMLから画像出力できる。

複数sampleを比較するなら、Multi-sample BAM QCから開始する。PCA plotなどが追加される。データも重ねて表示される(12sample例)。

リンク先から色々なデータを確認できます。
http://qualimap.bioinfo.cipf.es/doc_html/samples.html
引用
Qualimap 2: advanced multi-sample quality control for high-throughput sequencing data.
Okonechnikov K, Conesa A, García-Alcalde F.
Bioinformatics. 2016 Jan 15;32(2):292-4.
Qualimap: evaluating next-generation sequencing alignment data.
García-Alcalde F1, Okonechnikov K, Carbonell J, Cruz LM, Götz S, Tarazona S, Dopazo J, Meyer TF, Conesa A.
Bioinformatics. 2012 Oct 15;28(20):2678-9.