2018 10/30 イントロ修正
PanPhlAnはメタゲノムをstrainレベルで解析するツール。調べるのは遺伝子の有り/無しで、データベースのゲノムと比較することでメタゲムシーケンスしたバクテリアの特定の種に、実際にはどれくらいの多様性があるか(どれくらいのstrainが混じっているか)を調べることが可能となっている(本手法でプロファイルの指標になるのはコード領域の配列)。
PanPhlAnの解析例として、公式ページにはアウトブレイクしたE.coliのシーケンスデータをPanPhlAnで解析し、ヒートマップに可視化した図が載っている(リンク先の上の図a)。図1の下の方にある黒い線がシーケンスされ検出された菌ゲノムで、黄色がPanphlanのデータベースのゲノムである。データベースにはこれまでシーケンス解読され登録されてきた様々なE.coliのstrainが載っているのだが、その中でアウトブレイクしたE.coliの株は密接してクラスターを作っていることがわかる。これはアウトブレイクを起こしたE.coliのstrainのゲノムが互いに非常によく似ていることを意味する。
バクテリアのGWAS解析などができないかと妄想してこのツールを見つけたが、PanPhlAnは今後のメタゲノム解析に有用なツールと感じた。導入して使い勝手を見ていく。
公式サイト
https://bitbucket.org/CibioCM/panphlan/wiki/Home
https://bitbucket.org/CibioCM/panphlan/wiki/Tutorial
インストール
依存
- Python version 2.7 or 3.x (including the Biopython module, numpy, and matplotlib)
- bowtie2 #2.21リンク
- samtools
- usearch 7 #ダウンロード
Pythonとusearch以外はbrewでインストール可能。またpythonのモジュールはpipでインストールする。最後のusearch7はリンクよりダウンロードし、実行権をつけた上でusearch7という名前に変えてパスを通す。
本体のダウンロード
wget https://bitbucket.org/cibiocm/panphlan/get/default.zip
unzip default.zip
mkdir panphlan
mv CibioCM-panphlan-93907843bbfa/ panphlan/ #名前を変更しておく
プログラムが認識できる様に、以下の名前でパスを通しておく。
echo 'export PATH=/Users/user/local/bowtie2-2.2.1/:$PATH' >> ~/.bash_profile
echo 'export BT2_HOME=/Users/user/local/bowtie2-2.2.1/' >> ~/.bash_profile
echo 'export BOWTIE2_INDEXES=/Users/user/local/panphlan/Pre-processed_databases/' >> ~/.bash_profile
#ここでは書いていないがsamtoolsもパスが通っていなければ通す。
入力ファイル
fasta、fastq、tar.bz2、gzなど。
実行方法
以下の様な流れでランする。
データベースの作成
↓
シーケンスデータをデータベースの配列にアライメントする。
↓
結果をマージして1つにする。
↓
遺伝子セットの有無をまとめた行列データが出力される。
マニュアルに沿って実際にやってみる。
1、データベースのダウンロード。
wget http://www.matthias-scholz.de/panphlan_erectale15.zip
unzip panphlan_erectale15.zip
終わるとカレントディレクトリに8つのファイル panphlan_erectale15~ができる。このうち4つはbowtie2のindexファイルで、残りはPanPhlAnのデータベースファイルである。これらのファイルをデータベースとして使って以降の作業を行う。
2、シーケンスデータの準備。テストデータをダウンロードする。
wget -P samples https://www.dropbox.com/sh/5vxicumnz3adiwi/AACyBh4CUJFJ1P6a-ypzHvlua/SRS013951.tar.bz2
wget -P samples https://www.dropbox.com/sh/5vxicumnz3adiwi/AAA4CVLuQVzg9-9ql4pAQxL0a/SRS014459.tar.bz2
wget -P samples https://www.dropbox.com/sh/5vxicumnz3adiwi/AABslonWx4_V7L9ugEbSN3XFa/SRS015065.tar.bz
wget -P samples https://www.dropbox.com/sh/5vxicumnz3adiwi/AACanDqRyhaX0G6Dtf4NPTVwa/SRS019161.tar.bz2
4つ全て5GB以上あるので、空き容量に注意。
4ファイルできる。
ls samples/
SRS013951.tar.bz2 SRS014459.tar.bz2 SRS015065.tar.bz2 SRS019161.tar.bz2
シーケンスデータをデータベースの配列にマッピングする。
./panphlan/panphlan_map.py -c erectale15 -i samples/SRS013951.tar.bz2 -o map_results/SRS013951_erectale15.csv --verbose
./panphlan/panphlan_map.py -c erectale15 -i samples/SRS014459.tar.bz2 -o map_results/SRS014459_erectale15.csv --verbose
./panphlan/panphlan_map.py -c erectale15 -i samples/SRS015065.tar.bz2 -o map_results/SRS015065_erectale15.csv --verbose
./panphlan/panphlan_map.py -c erectale15 -i samples/SRS019161.tar.bz2 -o map_results/SRS019161_erectale15.csv --verbose
ダウンロードしたファイル4つそれぞれに対して実行する。
map_results/に4つのファイルができる。
4ファイルを付属のツールでまとめ、検出された遺伝子がどの種に存在するのかマトリックスにして出力する。
./panphlan/panphlan_profile.py -c erectale15 -i map_results/ --o_dna result_gene_presence_absence.csv --add_strains --verbose
ディレクトリを指令するだけで認識する。
ヒートマップを書く。
hclust2をダウンロード(ない人だけ)。
hg clone https://bitbucket.org/nsegata/hclust2
python2.7 hclust2/hclust2.py -i result_gene_presence_absence.csv -o heatmap.png --legend_file legend.png --image_size 30 --cell_aspect_ratio 0.0005 --dpi 300 --slabel_size 16 --f_dist_f jaccard --s_dist_f jaccard
図が出力される。

データベースを自分で作る場合。(リンク)
NCBIの ftp://ftp.ncbi.nlm.nih.gov/genomes/genbank/bacteria/ からデータを取ってくる。
wget -P fna ftp://ftp.ncbi.nih.gov/genomes/archive/old_refseq/Bacteria/Eubacterium_rectale_ATCC_33656_uid59169/NC_012781.fna
wget -P ffn ftp://ftp.ncbi.nih.gov/genomes/archive/old_refseq/Bacteria/Eubacterium_rectale_ATCC_33656_uid59169/NC_012781.ffn
wget -P fna ftp://ftp.ncbi.nih.gov/genomes/archive/old_refseq/Bacteria/Eubacterium_rectale_uid197161/NC_021010.fna
wget -P ffn ftp://ftp.ncbi.nih.gov/genomes/archive/old_refseq/Bacteria/Eubacterium_rectale_uid197161/NC_021010.ffn
wget -P fna ftp://ftp.ncbi.nih.gov/genomes/archive/old_refseq/Bacteria/Eubacterium_rectale_uid197162/NC_021044.fna
wget -P ffn ftp://ftp.ncbi.nih.gov/genomes/archive/old_refseq/Bacteria/Eubacterium_rectale_uid197162/NC_021044.ffn
3生物のfnaとffnが保存される。ffnはコード領域のみのfna。
データベースを作成。
sudo python2.7 panphlan_pangenome_generation.py -c erectale15 --i_fna fna/ --i_ffn ffn/ -o database/
- -c to specify the clade or species database-name;
- -i_fna input folder for genome sequences
- -i_fnn input folder for gene sequences
- -o output folder for the pangenome database
-cで指定した名前が以降使うデータベース名になる。ここでは"erectale15"。ランには数十分かかる。
データベース作成が終わったら、出力をチェック
user$ ls -l database/
total 57344
-rw-r--r-- 1 root staff 7622462 8 7 16:35 panphlan_erectale15.1.bt2
-rw-r--r-- 1 root staff 2568828 8 7 16:35 panphlan_erectale15.2.bt2
-rw-r--r-- 1 root staff 1979 8 7 16:35 panphlan_erectale15.3.bt2
-rw-r--r-- 1 root staff 2568822 8 7 16:35 panphlan_erectale15.4.bt2
-rw-r--r-- 1 root staff 7622462 8 7 16:35 panphlan_erectale15.rev.1.bt2
-rw-r--r-- 1 root staff 2568828 8 7 16:35 panphlan_erectale15.rev.2.bt2
-rw-r--r-- 1 root staff 5231770 8 7 16:35 panphlan_erectale15_centroids.ffn
-rw-r--r-- 1 root staff 1159076 8 7 16:35 panphlan_erectale15_pangenome.csv
database/に移動する。
データベースを調べるなら以下のコマンドを打つ。
sudo python2.7 ../panphlan/panphlan_profile.py -c erectale15 --add_strains --o_dna genefamily_presence_absence.tsv
出力ファイルは以下のようなファイルになっている。
user$ head genefamily_presence_absence.tsv
REF_NC_012781 REF_NC_021010 REF_NC_021044
g000001 1 0 0
g000002 1 0 0
g000003 1 0 0
g000004 1 0 0
g000005 1 0 0
g000006 1 0 0
g000007 1 0 0
g000008 1 0 0
g000009 1 0 0
データベースを作成した3株の列それぞれに、マーガーのヒット結果が1行ずつプリントされている。
プリセットデータベースをダウンロードする場合。(リンク)
hg clone https://bitbucket.org/CibioCM/panphlan
source panphlan/tools/download_all_databases.sh
mkdir Pre-processed_databases #フォルダを作成して
mv *zip Pre-processed_databases/ #その中にzipを入れる。
cd Pre-processed_databases/
unzip '*zip' #解凍。シングルクオーテーションで囲まないとエラーになる。
#元のデータを消す。残してもOK
rm Pre-processed_databases/*zip
hgがない人はbrewでhgコマンドを導入可能(ゲノムデータベースを自分で作る場合は一番下を参照)。合計14GBある。
解凍が終わると大量のファイルができる。それぞれのデータベースの、ffn、fna、bowtie2のindexファイル、またツール専用のファイルなどになる。
この様な感じで、Pre-processed_databases/には大量のファイルができている。
user$ ls -l Pre-processed_databases/panphlan_* |head -20
-rw-rw-rw- 1 user staff 6498035 3 6 2016 Pre-processed_databases/panphlan_aaphrophilus16.1.bt2
-rw-rw-rw- 1 user staff 1723384 3 6 2016 Pre-processed_databases/panphlan_aaphrophilus16.2.bt2
-rw-rw-rw- 1 user staff 458 3 6 2016 Pre-processed_databases/panphlan_aaphrophilus16.3.bt2
-rw-rw-rw- 1 user staff 1723376 3 6 2016 Pre-processed_databases/panphlan_aaphrophilus16.4.bt2
-rw-rw-rw- 1 user staff 6498035 3 6 2016 Pre-processed_databases/panphlan_aaphrophilus16.rev.1.bt2
-rw-rw-rw- 1 user staff 1723384 3 6 2016 Pre-processed_databases/panphlan_aaphrophilus16.rev.2.bt2
-rw-rw-rw- 1 user staff 2681949 3 6 2016 Pre-processed_databases/panphlan_aaphrophilus16_centroids.ffn
-rw-rw-rw- 1 user staff 577364 3 6 2016 Pre-processed_databases/panphlan_aaphrophilus16_pangenome.csv
-rw-rw-rw- 1 user staff 5963263 3 6 2016 Pre-processed_databases/panphlan_abirgiae16.1.bt2
-rw-rw-rw- 1 user staff 1325616 3 6 2016 Pre-processed_databases/panphlan_abirgiae16.2.bt2
-rw-rw-rw- 1 user staff 620 3 6 2016 Pre-processed_databases/panphlan_abirgiae16.3.bt2
-rw-rw-rw- 1 user staff 1325612 3 6 2016 Pre-processed_databases/panphlan_abirgiae16.4.bt2
-rw-rw-rw- 1 user staff 5963263 3 6 2016 Pre-processed_databases/panphlan_abirgiae16.rev.1.bt2
-rw-rw-rw- 1 user staff 1325616 3 6 2016 Pre-processed_databases/panphlan_abirgiae16.rev.2.bt2
-rw-rw-rw- 1 user staff 4911052 3 6 2016 Pre-processed_databases/panphlan_abirgiae16_centroids.ffn
-rw-rw-rw- 1 user staff 439546 3 6 2016 Pre-processed_databases/panphlan_abirgiae16_pangenome.csv
-rw-rw-rw- 1 user staff 4925884 3 6 2016 Pre-processed_databases/panphlan_acardiffensis16.1.bt2
-rw-rw-rw- 1 user staff 547012 3 6 2016 Pre-processed_databases/panphlan_acardiffensis16.2.bt2
-rw-rw-rw- 1 user staff 368 3 6 2016 Pre-processed_databases/panphlan_acardiffensis16.3.bt2
-rw-rw-rw- 1 user staff 547005 3 6 2016 Pre-processed_databases/panphlan_acardiffensis16.4.bt2
合計3341ファイル。
例えばaaphrophilusを解析するなら、上に出てくるaaphrophilus16をデータベース名として使う。
データベースが充実したecoli16を確認してみる。
cd Pre-processed_databases/
sudo chmod u+rx *
sudo python2.7 ../panphlan/panphlan_profile.py -c aaphrophilus16 -i map_results/ --o_dna result_gene_presence_absence.csv --add_strains
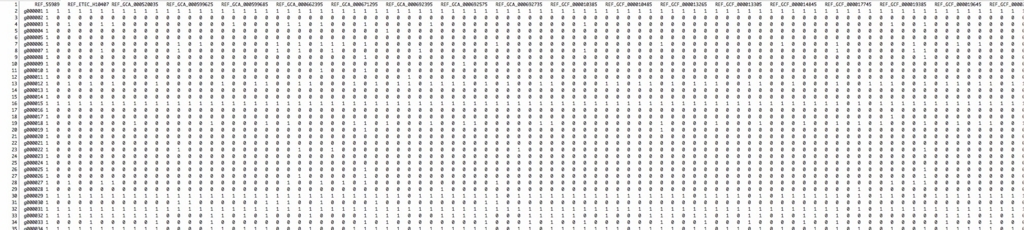
100以上あるE.coliのstrain(列)の遺伝子(行)の存在する/存在しないが1/0でマトリックスに表記されている。
他にも下のような解析が可能である。公式リンクを貼っておく。
→ RNA-seq: How to extract strain specific transcriptional activities?
→ Functional analysis: How to get gene-family sequences and annotations?
→ How to adapt strain detection thresholds?
gene familyのIDを指定して配列を取り出すやり方など参考になりそうである。
引用
Strain-level microbial epidemiology and population genomics from shotgun metagenomics.
Matthias Scholz*, Doyle V. Ward*, Edoardo Pasolli*, Thomas Tolio, Moreno Zolfo, Francesco Asnicar, Duy Tin Truong, Adrian Tett, Ardythe L. Morrow, and Nicola Segata (* Equal contribution)
Nature Methods, 13, 435–438, 2016.