2018 10/23 追記
2019 1/16 追記5
2019 3/16 bioconda追記6、canu -hヘルプ追記
2019 3/18 追記7
2019 4/12 追記8 dcokerリンク追加
2019 5/14 追記8リンク追加
2019 8/4 説明追加
2019 8/14 リンク追加
2019 11/5 canu v1.9 updateのお知らせtwitter追加
2019 11/11 biocondaインストールリンク追加、dockerを使ったrrun例を修正
2020 9/5 2.1 ツイート追記
1分子ロングリードシークエンシングはデノボゲノムアセンブリに革命をもたらし、参照品質ゲノムの自動再構成を可能にした。しかしながら、そのような技術の比較的高いエラー率を考慮すると、大きなリピート配列および密接に関連するハプロタイプの効率的かつ正確なアセンブルは依然として困難である。これらの問題は、ノイズの多い単一分子シーケンス用に特別に設計されたCelera Assemblerの後継製品であるCanuで解決する。 Canuはナノポアシークエンシングをサポートし、カバレッジ深度の要件を半分にし、アセンブリのcontiguityを向上させると同時に、Celera Assembler 8.2と比較して大きなゲノムでランタイムを一桁も短縮する。これらの進歩は tf-idf weighted MinHash と sparse assembly graph constructionによってリピートやハプロタイプによる崩壊を防ぐアセンブリアルゴリズムの結果である。著者らはCanuがPacific Biosciences(PacBio)またはOxford Nanoporeのいずれかを用いて完全なバクテリアゲノムおよびほぼ完全な真核生物染色体を確実にアセンブルし、ヒトおよびショウジョウバエのPacBioデータセットで> 21MbpのコンティグNG50を達成することを示す。
CanuはPacbioやnanoporeなどの1分子シーケンス用のアセンブラとして開発された。下記にはCanuを使ってヒトゲノムのアセンブリを行った例が紹介されている。
canuはPBcRの後継にもなっており、PBcRのダウンロードページでは、特別な理由がない限りPBcRの代わりにcanuをロングリードのアセンブリに使うことを推奨している。
http://wgs-assembler.sourceforge.net/wiki/index.php/PBcR
We are marking the occasion with a Canu v2.1 release. Update recommended for both both @PacBio and @nanopore assemblies, 5mo of fixes, check release notes https://t.co/vbSjYpvT0g
— Adam Phillippy (@aphillippy) 2020年9月1日
Canu v1.9 released 🎉 Includes support for @PacBio HiFi reads and fixes a number of embarrassing bugs discovered by letting @sergeynurk poke at the code for a few months. Update recommended! Release notes: https://t.co/agPz6lGoJE
— Adam Phillippy (@aphillippy) November 5, 2019
インストール
macはbinaryのみ提供されている。version1.5のページのcanu-1.5.Darwin-amd64.tar.xzをダウンロードした(mac OSXはDarwin系列のunix)。ダウンロードが終わったら解凍する。
gunzip -dc canu-1.5.Darwin-amd64.tar.xz | tar -xf -
できたディレクトリを適切な場所に移動させる。
ディレクトリ中Darwin-amd64/binに本体が入っているので、そこにパスを通せばランの準備は整う。
追記6
#bioconda(link)2019年11月現在1.9が最新
conda install -y -c bioconda canu
dockerhubにdockerイメージもたくさん上げられている。
https://hub.docker.com/search/?isAutomated=0&isOfficial=0&page=1&pullCount=0&q=canu&starCount=0
ここではbiocontainerのイメージを使う(tagリンク)。
docker pull biocontainers/canu:v1.8dfsg-2-deb_cv1
#カレントにデータがあるなら以下のような感じでランできる。ONTのデータ
docker run --rm -itv $PWD:/data/ -w /data biocontainers/canu:v1.8dfsg-2-deb_cv1 \
canu -p test -d assembly-directory genomeSize=4.8m -nanopore-raw oxford.fasta
> canu
$ canu
usage: canu [-correct | -trim | -assemble | -trim-assemble] \
[-s <assembly-specifications-file>] \
-p <assembly-prefix> \
-d <assembly-directory> \
genomeSize=<number>[g|m|k] \
errorRate=0.X \
[other-options] \
[-pacbio-raw | -pacbio-corrected | -nanopore-raw | -nanopore-corrected] *fastq
By default, all three stages (correct, trim, assemble) are computed.
To compute only a single stage, use:
-correct - generate corrected reads
-trim - generate trimmed reads
-assemble - generate an assembly
-trim-assemble - generate trimmed reads and then assemble them
The assembly is computed in the (created) -d <assembly-directory>, with most
files named using the -p <assembly-prefix>.
The genome size is your best guess of the genome size of what is being assembled.
It is used mostly to compute coverage in reads. Fractional values are allowed: '4.7m'
is the same as '4700k' and '4700000'
The errorRate is not used correctly (we're working on it). Don't set it
If you want to change the defaults, use the various utg*ErrorRate options.
A full list of options can be printed with '-options'. All options
can be supplied in an optional sepc file.
Reads can be either FASTA or FASTQ format, uncompressed, or compressed
with gz, bz2 or xz. Reads are specified by the technology they were
generated with:
-pacbio-raw <files>
-pacbio-corrected <files>
-nanopore-raw <files>
-nanopore-corrected <files>
Complete documentation at http://canu.readthedocs.org/en/latest/
ERROR: Assembly name prefix not supplied with -p.
ERROR: Directory not supplied with -d.
ERROR: Invalid 'corErrorRate' specified; must be set
ERROR: Required parameter 'genomeSize' is not set
追記
canu-racon dockerイメージ
https://hub.docker.com/r/staphb/canu-racon/
テストラン
公式ページで提供されているnanoporeのシーケンスデータ(fastaに変換されている)を使って、テストランを行う。
curl -L -o oxford.fasta http://nanopore.s3.climb.ac.uk/MAP006-PCR-1_2D_pass.fasta
oxford.fasta (145MB) がカレントディレクトリにダウンロードされる。
canu -p ecoli -d assembly-directory genomeSize=4.8m -nanopore-raw oxford.fasta
プリフィックスとしてecoliという名前をつけている。-dでランディレクトリを指定する。今回だとassembly-directoryディレクトリの中で、アセンブル前のエラーコレクション、エラーのトリミング、unitig作成が行われる。
ランが進むと、作業ディレクトリにレポートが出力される。
correction.htmlを開く。
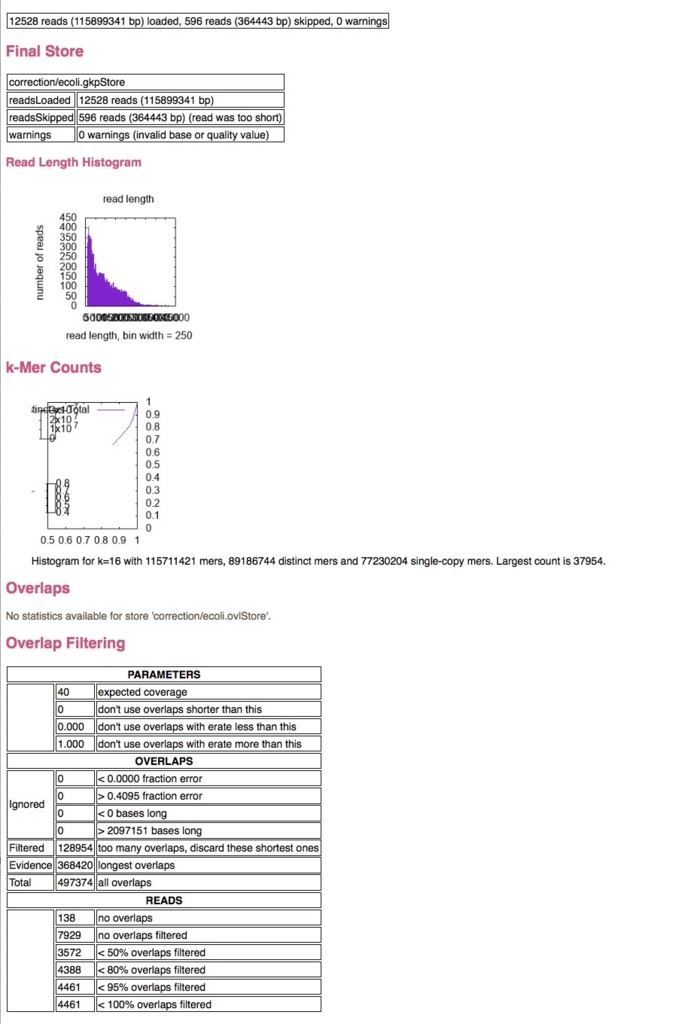
correction.html.files/中にはRで描かれたと思われるオリジナルのjpgが保存されている。インサートサイズのjpgを開く。

ポアソン分布に近い裾野が長い分布になっている。
ランが進むと、trimming.htmlができる。開くとトリミングに関するレポートをみることができる。
Pacbioのデータでも試してみる(Pacbioのテストラン)。
ダウンロード
curl -L -o p6.25x.fastq http://gembox.cbcb.umd.edu/mhap/raw/ecoli_p6_25x.filtered.fastq
p6.25x.fastq (233MB) がカレントディレクトリにダウンロードされる(fastqになっている)。
canu -p ecoli -d ecoli-auto genomeSize=4.8m -pacbio-raw p6.25x.fastq
エラーコクレションされたリード

エラーの多い1Dのデータをアセンブルすると、Miniasmだとできないなりに最後までプロセスは進みますが、canuはerrorで止まルようです。あまりにノイジーでアセンブルできないなら、ナノポアのロングリード自体でロングリードをpolishするか、illuminaのショートリードでポリッシュするようなフローが必要になります。ご注意ください。
以下の論文では5つのエラー修復方法を検討しています。
https://www.nature.com/articles/srep28625
追記1 2018/03時点ですでに100回以上引用されている(リンク)。中にはONTリードをcanuでアセンンブルして、ドラフトゲノムとしてgenome anouncementに出した論文も出てきている。。
canuは特にパラメータ設定次第でアセンブル結果がかなり変化するので、最適化するには複数回繰り返すのが望ましいでしょう。
追記4
Flyeというロングリードのアセンブラも出てきています。試した限り、canuより短時間かつ少ないリソースでアセンブリできました。アセンブリ精度についても、canuと同等か、それ以上になりました。ぜひcanuとFlye両方試してみてください。
追記5
canu 1.8に関するツイート
ロングリードのDNA配列をアセンブリするCanuのv1.8のdocker imege 作りましたのでお使い下さい.
— Yuji Yasuda (@yupeji) November 5, 2018
docker run -v $PWD:/data yupeji/canu:1.8 canu -option
Canu1.7 has a bug in building contigs. Version1.8 is preferred
— Andy Yuan (@Yuxuan_Yuan) January 14, 2019
追記7
canuの最適化
追記9
他のツールとの比較。
日本語だとこちらが参考になります。
引用
Canu: scalable and accurate long-read assembly via adaptive k-mer weighting and repeat separation.
Koren S, Walenz BP, Berlin K, Miller JR, Bergman NH, Phillippy AM
Genome Res. 2017 May;27(5):722-736.