全ゲノムシーケンス(WES)は、ヒトゲノムのタンパク質コーディングエクソンのターゲットシーケンスであり、新しいメンデル遺伝病遺伝子を特定するための強力で費用対効果の高い方法であり、診断環境でもますます使用されている[Bamshad et al 、2011; Robinson et al、2011; Shendure、2011; Choi et al、2012]。 2005年に次世代シーケンス(NGS)が導入されて以来[Margulies et al、2005; Shendure et al、2005]および2010年のWESによるメンデル病遺伝子の最初の同定[Ng et al、2010]、WESにより同定された100以上の新規疾患遺伝子が発表された[Rabbani et al、2012]。現在、WESのコストは1,000米ドルを下回り、急速に低下しているため、ヒトの遺伝学およびその他の医学分野の研究および臨床診断にWESを使用する新しい時代に突入している。
WESデータの生成は急速に容易かつ安価になっているが、これらのデータの分析と解釈は依然として課題である。使用するターゲット領域とキャプチャ技術の定義に応じて、典型的なWES実験で20,000を超えるバリアントが識別される[Ng et al、2009]。rawシーケンシングリードからのバリアントの同定には、リードのリファレンスゲノムへのマッピングや、1つ以上のアルゴリズムを使用したバリアントコールなど、多くの処理ステップが含まれる。この分析の結果はVariant Call Format(VCF)ファイルに保存される。このファイルには、特定された各バリアントの染色体位置、リードデプス、クオリティ、およびその他のメタデータに関する情報が含まれている[Danecek et al、2011]。このデータの解釈における重要なステップは、遺伝子および転写産物に対する潜在的な影響に関するこれらのバリアントのアノテーションである。つまり、染色体座標を反映するVCFファイルのバリアントを変換する(例、chr11:g.1857751C>G)、遺伝子ベースのバリアントアノテーションを行う(例:c.655C>G:p.P219A in the gene SYT8)。ほとんどの生物学的または医学的解釈は、遺伝子産物に対するバリアントの潜在的な影響を評価しようとする。
ANCF [Wang et al、2010]、クラウドコンピューティングフレームワークVAT [Habegger et al、2012]、Variant Effect PredictorなどVCFファイルおよびその他のソースからのゲノムバリエーションにアノテーションを付けるための多くのツールが開発されている。ただし、これらのツールは血統分析を実行するように設計されておらず、多くは5 'または3'非翻訳領域(UTR)のバリアントなどの特定のクラスのバリアントに正確なアノテーションを提供せず、ソフトウェアライブラリとして使用できない。 Jannovarは、高度なエクソームシーケンスソフトウェアパイプラインのバリアントアノテーションおよび血統分析のための柔軟で十分にテストされたソフトウェアライブラリのニーズを満たすために開発され、さらにVCFのアノテーションのための迅速で使いやすいスタンドアロンJavaプログラムを提供する。 JannovarはJavaプログラミング言語で作成されており、開発者はバリアントの解釈、視覚化、優先順位付け、および関連タスクのプログラムのコンポーネントとして使用できる。 Jannovarは、カリフォルニア大学サンタクルーズ(UCSC)ゲノムブラウザ[Meyer et al、2013]、NCBI RefSeq [Pruitt et al、2012]、またはEnsembl [Flicek et al、2013]データからトランスクリプト定義ファイルを作成する。迅速で信頼性の高いインターバルツリーベースのアルゴリズムを使用して、バリアントの影響を受ける転写産物を検索し、エキソン変異と5 'および3' UTRにある変異および非コードRNA変異のHuman Gene Variation Society(HGVS; Antonarakis、1998)compliant variant nomenclature を生成する。
Jannovarのソースコードは、GitHubリポジトリhttps://github.com/charite/jannovarから入手できる。プリコンパイルされたバージョン(Jannovar.jar)は、詳細なチュートリアルと共に、http://compbio.charite.deのホームページから入手できる。 Jannovarは、スタンドアロンアプリケーションとして使用する場合、エクソームまたはゲノムシーケンスからのVCFファイルのHGVS準拠の高速アノテーションを提供し、継承モードに従ってバリアントをフィルタリングできる。さらに、Jannovarは、エクソームフィルタリングのプログラムまたはパイプライン内でJavaプログラミングライブラリとして使用できる。(以下略)
インストール
依存
本体 Github
#bioconda (link)
conda install -c bioconda -y jannovar-cli
> jannovar
$ jannovar
usage: jannovar-cli [-h] [--version] {annotate-pos,annotate-csv,annotate-vcf,db-list,download,statistics,rest-server,hgvs-to-vcf} ...
jannovar-cli: error: too few arguments
(base) kamisakakazumanoMac-mini:deletion kazu$ jannovar -h
usage: jannovar-cli [-h] [--version] {annotate-pos,annotate-csv,annotate-vcf,db-list,download,statistics,rest-server,hgvs-to-vcf} ...
Jannovar CLI performs a series of VCF annotation tasks, including predicted molecular impact of variants and annotation of compatible Mendelian inheritance.
positional arguments:
{annotate-pos,annotate-csv,annotate-vcf,db-list,download,statistics,rest-server,hgvs-to-vcf}
annotate-pos annotate genomic changes given on the command line
annotate-csv Annotate a csv file
annotate-vcf annotate VCF files
db-list list databases available for download
download download transcript databases
statistics compute statistics about VCF file
rest-server start REST server
hgvs-to-vcf project transcript-level to chromosome-level changes
optional arguments:
-h, --help show this help message and exit
--version Show Jannovar version
You can find out more at http://jannovar.rtfd.org
> jannovar annotate-vcf -h
$ jannovar annotate-vcf -h
usage: jannovar-cli annotate-vcf [-h] -i INPUT_VCF -o OUTPUT_VCF -d DATABASE [--interval INTERVAL] [--pedigree-file PEDIGREE_FILE] [--annotate-as-singleton-pedigree] [--ref-fasta REF_FASTA] [--dbsnp-vcf DBSNP_VCF] [--dbsnp-prefix DBSNP_PREFIX]
[--exac-vcf EXAC_VCF] [--exac-prefix EXAC_PREFIX] [--gnomad-exomes-vcf GNOMAD_EXOMES_VCF] [--gnomad-exomes-prefix GNOMAD_EXOMES_PREFIX] [--gnomad-genomes-vcf GNOMAD_GENOMES_VCF]
[--gnomad-genomes-prefix GNOMAD_GENOMES_PREFIX] [--uk10k-vcf UK10K_VCF] [--uk10k-prefix UK10K_PREFIX] [--g1k-vcf G1K_VCF] [--g1k-prefix G1K_PREFIX] [--clinvar-vcf CLINVAR_VCF] [--clinvar-prefix CLINVAR_PREFIX]
[--cosmic-vcf COSMIC_VCF] [--cosmic-prefix COSMIC_PREFIX] [--one-parent-gt-filtered-filters-affected] [--inheritance-anno-use-filters] [--dbnsfp-tsv DBNSFP_TSV] [--dbnsfp-col-contig DBNSFP_COL_CONTIG]
[--dbnsfp-col-position DBNSFP_COL_POSITION] [--dbnsfp-prefix DBNSFP_PREFIX] [--dbnsfp-columns DBNSFP_COLUMNS] [--bed-annotation BED_ANNOTATION] [--vcf-annotation VCF_ANNOTATION] [--tsv-annotation TSV_ANNOTATION]
[--use-threshold-filters] [--gt-thresh-filt-min-cov-het GT_THRESH_FILT_MIN_COV_HET] [--gt-thresh-filt-min-cov-hom-alt GT_THRESH_FILT_MIN_COV_HOM_ALT] [--gt-thresh-filt-max-cov GT_THRESH_FILT_MAX_COV]
[--gt-thresh-filt-min-gq GT_THRESH_FILT_MIN_GQ] [--gt-thresh-filt-min-aaf-het GT_THRESH_FILT_MIN_AAF_HET] [--gt-thresh-filt-max-aaf-het GT_THRESH_FILT_MAX_AAF_HET]
[--gt-thresh-filt-min-aaf-hom-alt GT_THRESH_FILT_MIN_AAF_HOM_ALT] [--gt-thresh-filt-max-aaf-hom-ref GT_THRESH_FILT_MAX_AAF_HOM_REF] [--var-thresh-max-allele-freq-ad VAR_THRESH_MAX_ALLELE_FREQ_AD]
[--var-thresh-max-allele-freq-ar VAR_THRESH_MAX_ALLELE_FREQ_AR] [--var-thresh-max-hom-alt-exac VAR_THRESH_MAX_HOM_ALT_EXAC] [--var-thresh-max-hom-alt-g1k VAR_THRESH_MAX_HOM_ALT_G1K] [--use-advanced-pedigree-filters]
[--de-novo-max-parent-ad2 DE_NOVO_MAX_PARENT_AD2] [--enable-off-target-filter] [--utr-is-off-target] [--intronic-splice-is-off-target] [--no-escape-ann-field] [--show-all] [--no-3-prime-shifting] [--3-letter-amino-acids]
[--disable-parent-gt-is-filtered] [--version] [--report-no-progress] [-v] [-vv] [--http-proxy HTTP_PROXY] [--https-proxy HTTPS_PROXY] [--ftp-proxy FTP_PROXY]
Perform annotation of a single VCF file
optional arguments:
-h, --help show this help message and exit
--version Show Jannovar version
Required arguments:
-i INPUT_VCF, --input-vcf INPUT_VCF
Path to input VCF file
-o OUTPUT_VCF, --output-vcf OUTPUT_VCF
Path to output VCF file
-d DATABASE, --database DATABASE
Path to database .ser file
--interval INTERVAL Interval with regions to annotate (optional)
Annotation Arguments (optional):
--pedigree-file PEDIGREE_FILE
Pedigree file to use for Mendelian inheritance annotation
--annotate-as-singleton-pedigree
Annotate VCF file with single individual as singleton pedigree (singleton assumed to be affected)
--ref-fasta REF_FASTA Path to FAI-indexed reference FASTA file, required for dbSNP/ExAC/UK10K-based annotation
--dbsnp-vcf DBSNP_VCF Path to dbSNP VCF file, activates dbSNP annotation
--dbsnp-prefix DBSNP_PREFIX
Prefix for dbSNP annotations
--exac-vcf EXAC_VCF Path to ExAC VCF file, activates ExAC annotation
--exac-prefix EXAC_PREFIX
Prefix for ExAC annotations
--gnomad-exomes-vcf GNOMAD_EXOMES_VCF
Path to gnomAD exomes VCF file, activates gnomAD exomes annotation
--gnomad-exomes-prefix GNOMAD_EXOMES_PREFIX
Prefix for ExgnomAD exomes AC annotations
--gnomad-genomes-vcf GNOMAD_GENOMES_VCF
Path to gnomAD genomes VCF file, activates gnomAD genomes annotation
--gnomad-genomes-prefix GNOMAD_GENOMES_PREFIX
Prefix for ExgnomAD genomes AC annotations
--uk10k-vcf UK10K_VCF Path to UK10K VCF file, activates UK10K annotation
--uk10k-prefix UK10K_PREFIX
Prefix for UK10K annotations
--g1k-vcf G1K_VCF Path to thousand genomes VCF file, activates thousand genomes annotation
--g1k-prefix G1K_PREFIX
Prefix for thousand genomes annotations
--clinvar-vcf CLINVAR_VCF
Path to ClinVar file, activates ClinVar annotation
--clinvar-prefix CLINVAR_PREFIX
Prefix for ClinVar annotations
--cosmic-vcf COSMIC_VCF
Path to COSMIC file, activates COSMIC annotation
--cosmic-prefix COSMIC_PREFIX
Prefix for COSMIC annotations
--one-parent-gt-filtered-filters-affected
If one parent's genotype is affected, apply OneParentGtFiltered filter to child
--inheritance-anno-use-filters
Use filters in inheritance mode annotation
Annotation with dbNSFP (experimental; optional):
--dbnsfp-tsv DBNSFP_TSV
Patht to dbNSFP TSV file
--dbnsfp-col-contig DBNSFP_COL_CONTIG
Column index of contig in dbNSFP
--dbnsfp-col-position DBNSFP_COL_POSITION
Column index of position in dbNSFP
--dbnsfp-prefix DBNSFP_PREFIX
Prefix for dbNSFP annotations
--dbnsfp-columns DBNSFP_COLUMNS
Columns from dbDSFP file to use for annotation
BED-based Annotation (experimental; optional):
--bed-annotation BED_ANNOTATION
Add BED file to use for annotating. The value must be of the format "pathToBed:infoField:description[:colNo]".
Generic VCF-based Annotation (experimental; optional):
--vcf-annotation VCF_ANNOTATION
Add VCF file to use for annotating. The value must be of the format "pathToVfFile:prefix:field1,field2,field3".
TSV-based Annotation (experimental; optional):
--tsv-annotation TSV_ANNOTATION
Add TSV file to use for annotating. The value must be of the format "pathToTsvFile:oneBasedOffset:colContig:colStart:colEnd:colRef(or=0):colAlt(or=0):isRefAnnotated(R=yes,A=no):colValue:fieldType:fieldName:
fieldDescription:accumulationStrategy".
Threshold-filter related arguments:
--use-threshold-filters
Use threshold-based filters
--gt-thresh-filt-min-cov-het GT_THRESH_FILT_MIN_COV_HET
Minimal coverage for het. call
--gt-thresh-filt-min-cov-hom-alt GT_THRESH_FILT_MIN_COV_HOM_ALT
Minimal coverage for hom. alt calls
--gt-thresh-filt-max-cov GT_THRESH_FILT_MAX_COV
Maximal coverage for a sample
--gt-thresh-filt-min-gq GT_THRESH_FILT_MIN_GQ
Minimal genotype call quality
--gt-thresh-filt-min-aaf-het GT_THRESH_FILT_MIN_AAF_HET
Minimal het. call alternate allele fraction
--gt-thresh-filt-max-aaf-het GT_THRESH_FILT_MAX_AAF_HET
Maximal het. call alternate allele fraction
--gt-thresh-filt-min-aaf-hom-alt GT_THRESH_FILT_MIN_AAF_HOM_ALT
Minimal hom. alt call alternate allele fraction
--gt-thresh-filt-max-aaf-hom-ref GT_THRESH_FILT_MAX_AAF_HOM_REF
Maximal hom. ref call alternate allele fraction
--var-thresh-max-allele-freq-ad VAR_THRESH_MAX_ALLELE_FREQ_AD
Maximal allele fraction for autosomal dominant inheritance mode
--var-thresh-max-allele-freq-ar VAR_THRESH_MAX_ALLELE_FREQ_AR
Maximal allele fraction for autosomal recessive inheritance mode
--var-thresh-max-hom-alt-exac VAR_THRESH_MAX_HOM_ALT_EXAC
Maximal count in homozygous state in ExAC before ignoring
--var-thresh-max-hom-alt-g1k VAR_THRESH_MAX_HOM_ALT_G1K
Maximal count in homozygous state in ExAC before ignoring
--use-advanced-pedigree-filters
Use advanced pedigree-based filters (mainly useful for de novo variants)
--de-novo-max-parent-ad2 DE_NOVO_MAX_PARENT_AD2
Maximal support of alternative allele in parent for de novo variants.
Exome on/off target filters:
--enable-off-target-filter
Enable filter for on/off-target based on effect impact
--utr-is-off-target Make UTR count as off-target (default is to count UTR as on-target)
--intronic-splice-is-off-target
Make intronic (non-consensus site) splice region count as off-target (default is to count as on-target)
Other, optional Arguments:
--no-escape-ann-field Disable escaping of INFO/ANN field in VCF output
--show-all Show all effects
--no-3-prime-shifting Disable shifting towards 3' of transcript
--3-letter-amino-acids
Enable usage of 3 letter amino acid codes
--disable-parent-gt-is-filtered
Verbosity Options:
--report-no-progress Disable progress report, more quiet mode
-v, --verbose Enable verbose mode
-vv, --very-verbose Enable very verbose mode
Proxy Options:
Configuration related to Proxy, note that environment variables *_proxy and *_PROXY are also interpreted
--http-proxy HTTP_PROXY
Set HTTP proxy to use, if any
--https-proxy HTTPS_PROXY
Set HTTPS proxy to use, if any
--ftp-proxy FTP_PROXY Set FTP proxy to use, if any
> jannovar annotate-csv -h
$ jannovar annotate-csv -h
usage: jannovar-cli annotate-csv [-h] -d DATABASE -i INPUT -c CHR -p POS -r REF -a ALT [-t {Default,TDF,RFC4180,Excel,MySQL}] [--header] [--show-all] [--no-3-prime-shifting] [--3-letter-amino-acids] [--version] [--report-no-progress] [-v] [-vv]
[--http-proxy HTTP_PROXY] [--https-proxy HTTPS_PROXY] [--ftp-proxy FTP_PROXY]
Perform annotation of genomic changes given on the command line
optional arguments:
-h, --help show this help message and exit
--version Show Jannovar version
Required arguments:
-d DATABASE, --database DATABASE
Path to database .ser file
-i INPUT, --input INPUT
CSV file
-c CHR, --chr CHR Column of chr (1 based)
-p POS, --pos POS Column of pos (1 based)
-r REF, --ref REF Column of ref (1 based)
-a ALT, --alt ALT Column of alt (1 based)
Additional CSV arguments (optional):
-t {Default,TDF,RFC4180,Excel,MySQL}, --type {Default,TDF,RFC4180,Excel,MySQL}
Type of csv file.
--header Set if the file contains a header.
Optional Arguments:
--show-all Show all effects
--no-3-prime-shifting Disable shifting towards 3' of transcript
--3-letter-amino-acids
Enable usage of 3 letter amino acid codes
Verbosity Options:
--report-no-progress Disable progress report, more quiet mode
-v, --verbose Enable verbose mode
-vv, --very-verbose Enable very verbose mode
Proxy Options:
Configuration related to Proxy, note that environment variables *_proxy and *_PROXY are also interpreted
--http-proxy HTTP_PROXY
Set HTTP proxy to use, if any
--https-proxy HTTPS_PROXY
Set HTTPS proxy to use, if any
--ftp-proxy FTP_PROXY Set FTP proxy to use, if any
Example: java -jar Jannovar.jar annotate-csv -d hg19_refseq.ser -c 1 -p 2 -r 3 -r 4 -t TDF --header -i input.csv
> jannovar statistics -h
$ jannovar statistics -h
usage: jannovar-cli statistics [-h] -i INPUT_VCF -o OUTPUT_REPORT -d DATABASE [--version] [--report-no-progress] [-v] [-vv] [--http-proxy HTTP_PROXY] [--https-proxy HTTPS_PROXY] [--ftp-proxy FTP_PROXY]
Compute statistics about variants in VCF file
optional arguments:
-h, --help show this help message and exit
--version Show Jannovar version
Required arguments:
-i INPUT_VCF, --input-vcf INPUT_VCF
Path to input VCF file
-o OUTPUT_REPORT, --output-report OUTPUT_REPORT
Path to output report TXT file
-d DATABASE, --database DATABASE
Path to database .ser file
Verbosity Options:
--report-no-progress Disable progress report, more quiet mode
-v, --verbose Enable verbose mode
-vv, --very-verbose Enable very verbose mode
Proxy Options:
Configuration related to Proxy, note that environment variables *_proxy and *_PROXY are also interpreted
--http-proxy HTTP_PROXY
Set HTTP proxy to use, if any
--https-proxy HTTPS_PROXY
Set HTTPS proxy to use, if any
--ftp-proxy FTP_PROXY Set FTP proxy to use, if any
> jannovar annotate-pos -h
$ jannovar annotate-pos -h
usage: jannovar-cli annotate-pos [-h] -d DATABASE -c GENOMIC_CHANGE [--show-all] [--no-3-prime-shifting] [--3-letter-amino-acids] [--version] [--report-no-progress] [-v] [-vv] [--http-proxy HTTP_PROXY] [--https-proxy HTTPS_PROXY]
[--ftp-proxy FTP_PROXY]
Perform annotation of genomic changes given on the command line
optional arguments:
-h, --help show this help message and exit
--version Show Jannovar version
Required arguments:
-d DATABASE, --database DATABASE
Path to database .ser file
-c GENOMIC_CHANGE, --genomic-change GENOMIC_CHANGE
Genomic change to annotate, you can give multiple ones
Optional Arguments:
--show-all Show all effects
--no-3-prime-shifting Disable shifting towards 3' of transcript
--3-letter-amino-acids
Enable usage of 3 letter amino acid codes
Verbosity Options:
--report-no-progress Disable progress report, more quiet mode
-v, --verbose Enable verbose mode
-vv, --very-verbose Enable very verbose mode
Proxy Options:
Configuration related to Proxy, note that environment variables *_proxy and *_PROXY are also interpreted
--http-proxy HTTP_PROXY
Set HTTP proxy to use, if any
--https-proxy HTTPS_PROXY
Set HTTPS proxy to use, if any
--ftp-proxy FTP_PROXY Set FTP proxy to use, if any
Example: java -jar Jannovar.jar annotate-pos -d hg19_refseq.ser -c 'chr1:12345C>A'
> jannovar download -h
$ jannovar download -h
usage: jannovar-cli download [-h] -d DATABASE [-s DATA_SOURCE_LIST] [--download-dir DOWNLOAD_DIR] [--gene-ids GENE_IDS [GENE_IDS ...]] [-o OUTPUT_FILE] [--version] [--report-no-progress] [-v] [-vv] [--http-proxy HTTP_PROXY]
[--https-proxy HTTPS_PROXY] [--ftp-proxy FTP_PROXY]
Download transcript database
optional arguments:
-h, --help show this help message and exit
--version Show Jannovar version
Required arguments:
-d DATABASE, --database DATABASE
Name of database to download, can be given multiple times
Optional Arguments:
-s DATA_SOURCE_LIST, --data-source-list DATA_SOURCE_LIST
INI file with data source list
--download-dir DOWNLOAD_DIR
Path to download directory
--gene-ids GENE_IDS [GENE_IDS ...]
Optional list of genes to limit creation of database to
-o OUTPUT_FILE, --output-file OUTPUT_FILE
Optional path to output file
Verbosity Options:
--report-no-progress Disable progress report, more quiet mode
-v, --verbose Enable verbose mode
-vv, --very-verbose Enable very verbose mode
Proxy Options:
Configuration related to Proxy, note that environment variables *_proxy and *_PROXY are also interpreted
--http-proxy HTTP_PROXY
Set HTTP proxy to use, if any
--https-proxy HTTPS_PROXY
Set HTTPS proxy to use, if any
--ftp-proxy FTP_PROXY Set FTP proxy to use, if any
実行方法
1、データベースの準備(初回のみ)
hg19/GRCh37のRefSeq transcript databaseをダウンロードする。
jannovar download -d hg19/refseq
ダウンロード後、データベースファイルdata/hg19_refseq.serができる。

以下のデータベースが利用できる。
> jannovar db-list
$ jannovar db-list
Options
JannovarDBOptions [dataSourceFiles=[bundle:///default_sources.ini], isReportProgress()=true, getHttpProxy()=null, getHttpsProxy()=null, getFtpProxy()=null]
Available data sources:
hg18/ucsc
hg18/ensembl
hg18/refseq
hg18/refseq_curated
hg19/ucsc
hg19/ensembl
hg19/refseq
hg19/refseq_curated
hg19/refseq_interim
hg19/refseq_interim_curated
hg38/ucsc
hg38/ensembl
hg38/refseq
hg38/refseq_curated
mm9/ucsc
mm9/ensembl
mm9/refseq
mm9/refseq_curated
mm10/ucsc
mm10/ensembl
mm10/refseq
mm10/refseq_curated
rn6/refseq
rn6/refseq_curated
利用できるのはヒトとマウスのhg18, hg19, hg38, mm9, mm10である。
2、vcfを指定してバリアントのアノテーションを行う。
変異のVCFを指定する。ここではJannovarのgithubにあるsmall.vcfを使う。
jannovar annotate-vcf -d data/hg19_refseq.ser \
-i jannovar/examples/small.vcf -o ouput.vcf
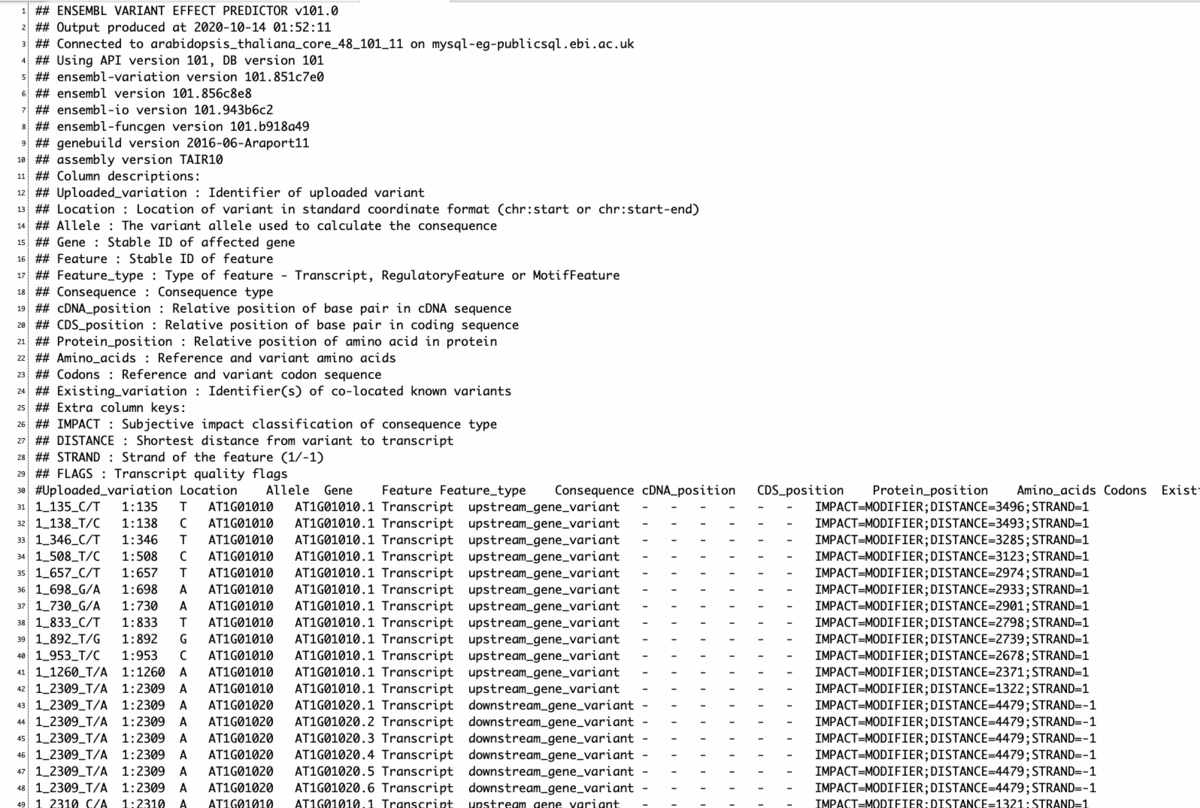
入力VCF

出力VCF。アノテーションがアサインされている。

"LOW"とか"MODERATE"などあるが、これはVariant annotations in VCF formatの定義に従って変異の影響度がつけられたものになる。
Variant Effects — Jannovar 0.11.0 documentation にも解説がある。

そのほかのコマンド
・jannovar annotate-pos - Perform annotation of genomic changes given on the command line
変異後の配列のアノテーションを素早く確認する。chr1の12345のC =>Aなら'chr1:12345C>A'と指定する(formatは{CHROMOSOME}:{POSITION}{REF}>{ALT} )。下ではもう1箇所指定している。
jannovar annotate-vcf -d data/hg19_refseq.ser \
-c 'chr1:12345C>A' -c 'chr1:12346C>A'
出力
#change effect hgvs_annotation messages
chr1:12345C>A NON_CODING_TRANSCRIPT_INTRON_VARIANT DDX11L1:NR_046018.2:n.354+118C>A: .
chr1:12346C>A NON_CODING_TRANSCRIPT_INTRON_VARIANT DDX11L1:NR_046018.2:n.354+119C>A: .
・jannovar statistics - Compute statistics about variants in VCF file
jannovar statistics -d data/hg19_refseq.ser \
-i input.vcf -o stats
出力

引用
Jannovar: a java library for exome annotation
Jäger M1, Wang K, Bauer S, Smedley D, Krawitz P, Robinson PN
Hum Mutat. 2014 May;35(5):548-55