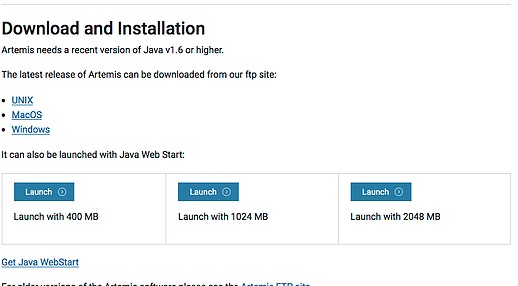
banファイルを読み込んでリード情報を可視化できるフリーソフト。多様なオプションが用意されており、使いこなすにはかなり勉強が必要。 例を挙げて説明する。
1、Split view 表示
下のようにウィンドウを分けて表示できる。本来はmate-pairの相方リード表示用だが、複数領域を同時に比較することにも利用できる。 やり方はMiなどのテキストエディタにあらかじめタブで区切った二つのポジションを書いておき、それをウィンドウ内にペーストするだけでOK。
例えばchrの471900-474900と519,700-522,701を同時にみたいなら、間をタブで区切り以下のように記載する。
chr:471900-474900 chr:519,700-522,701
二つの領域が表示される。以下は4つ表示した例。
split viewを解除するには、上のウィンドウの空間でダブルクリックする。
2、変異のコール結果のVCFファイルをIGVに取り込ませる
gatkの検出結果を読み込ませてみる。例えば
gatk -T UnifiedGenotyper -R $f -I R1R2_sorted.bam -o UnifiedGenotyper.vcf
出力されるvcfファイルを読み込ませる。

indel検出ソフトの出力はVCF形式に対応しているものが多い。変異をコールして気になる部位があればIGVで見てみればいい。主観なので危険性は伴うが、偽陽性かどうか推測できる場合も多い。
3、ゲノムのGC含量をIGVで表示。
例えば100bpのウィンドウサイズで描画したいとするなら、以下のように行う(1-bpのサイズは意味がないので、ある幅のウィンドウサイズで計算する)
解析に必要なもの
・samtools
・bed tools
(homebrewがない人は前もって導入しておく。ターミナルでbrewと打ってコマンドが見つからなければ入ってない)
gawkと同様にsamtoolsとbedtoolsの導入は
brew install samtools
brew install bedtools
ゲノム(test.faとする)のGC%を100bpのウィンドウサイズで計算してIGVで可視化するには、以下のようなフローで行なう。
1、fastaのindex作成
samtools test.fa
2、カラム1、2を抽出
cut -f 1,2 test.fasta.fai > test.sizes
3、GC計算幅を指定するファイルを作成
bedtools makewindows -g test.sizes -w 100 > test_100bps.bed
(w=100ならこんなのができる)
chr 0 100
chr 100 200
chr 200 300
chr 300 400
chr 400 500
….
4、ATGC分布を計算。
bedtools nuc -fi test.fasta -bed test_100bps.bed > test_nuc.txt
こんなのができる
head -5 test_100bps.bed
#1_usercol 2_usercol 3_usercol 4_pct_at 5_pct_gc 6_num_A 7_num_C 8_num_G 9_num_T 10_num_N 11_num_oth 12_seq_len
chr 0 100 0.390000 0.610000 20 25 36 19 0 0 100
chr 100 200 0.560000 0.440000 20 18 26 36 0 0 100
chr 200 300 0.580000 0.420000 31 15 27 27 0 0 100
chr 300 400 0.500000 0.500000 23 23 27 27 0 0 100
5、必要なカラムを編集しながら抽出。gawkを使っている。
gawk -v w=100 'BEGIN{FS=" "; OFS=" "}
{if (FNR>1) {print $1,$2,$3,"GCpc_"w"bps",$5}}' test_nuc.txt > test_100bp.igv
成功するとこんなファイルができる。
head -4 test_100bp.igv
chr 0 100 GCpc_100bps 0.610000
chr 100 200 GCpc_100bps 0.440000
chr 200 300 GCpc_100bps 0.420000
chr 300 400 GCpc_100bps 0.500000
6、test_100bp.igvをIGVに読み込ませる。
おまけ;
IGVtoolsでバイナリー化しておけば軽くなる。
IGVtools -> Run igvtools

Command -> toTDF を選択
input file Browse -> .igvファイルを選択。
Runをクリック

test_100bp.igvができる。Load from genomesから読み込ませる。igvtoolsは、この例のようにIGVのGUIから呼び出して計算させることができる。
--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------
参考ページ
http://wiki.bits.vib.be/index.php/Create_a_GC_content_track